22 Module Pandas¶
Le module pandas a été conçu pour l'analyse de données. Il est particulièrement puissant pour manipuler des données structurées sous forme de tableau.
22.1 Installation et convention¶
Le module pandas n'est pas fourni avec la distribution Python de base. Avec la distribution Miniconda que nous vous conseillons d'utiliser (consultez pour cela la documentation en ligne), vous pouvez rapidement l'installer avec la commande :
Vous aurez également besoin des modules matplotlib pour créer des graphiques et scipy pour réaliser une régression linaire, que vous pouvez installer ainsi :
Dans ce chapitre, nous vous montrerons quelques exemples d’utilisation du module pandas pour vous convaincre de sa pertinence. Ces exemples seront exécutés dans un notebook Jupyter.
Les cellules de code apparaitront de cette manière
dans un notebook Jupyter, avec des numéros de lignes à gauche.
22.2 Chargement du module¶
Pour charger pandas dans la mémoire de Python, on utilise la commande import habituelle :
Par convention, on utilise pd comme nom raccourci pour pandas :
22.3 Series¶
Le premier type de données apporté par pandas est la Series, qui correspond à un vecteur à une dimension.
22.3.1 Sélections par étiquette ou indice¶
Avec pandas, chaque élément de la série de données possède une étiquette qui permet d'appeler les éléments qui la composent.
Ainsi, pour appeler le premier élément de la série, on peut utiliser son étiquette (ici, "a") :
Pour accéder au premier élément par son indice (ici 0), comme on le ferait avec une liste, on utilise la méthode .iloc :
Bien sûr, on peut extraire plusieurs éléments, par leurs indices ou leurs étiquettes :
et
22.3.2 Modifications de Series¶
Les étiquettes permettent de modifier et d'ajouter des éléments :
22.3.3 Filtres¶
Enfin, on peut filtrer une partie de la Series :
Cette écriture rappelle celle des masques booléens dans le chapitre 20 Module NumPy.
Enfin, on peut aussi combiner plusieurs critères de sélection avec les opérateurs logiques & (pour ET) et | (pour OU) :
22.4 Dataframes¶
Un autre type d'objet particulièrement intéressant introduit par pandas sont les Dataframes. Ceux-ci correspondent à des tableaux à deux dimensions avec des étiquettes pour nommer les lignes et les colonnes.
Si vous êtes familier avec le langage de programmation et d'analyse statistique R, les Dataframes de pandas se rapprochent de ceux trouvés dans R.
22.4.1 Création¶
Voici comment créer un Dataframe avec pandas à partir de données fournies comme liste de lignes :
import numpy as np
df = pd.DataFrame(columns=["a", "b", "c", "d"],
index=["chat", "singe", "souris"],
data=[np.arange(10, 14),
np.arange(20, 24),
np.arange(30, 34)])
df
Voici quelques commentaires sur le code précédent :
- Ligne 1. On charge le module NumPy utilisé ensuite.
- Ligne 2. Le Dataframe est créé avec la fonction
DataFrame()à laquelle on fournit plusieurs arguments. L'argumentcolumnsindique le nom des colonnes, sous forme d'une liste. - Ligne 3. L'argument
indexdéfinit le nom des lignes, sous forme de liste également. - Lignes 4 à 6. L'argument
datafournit le contenu du Dataframe, sous la forme d'une liste de valeurs correspondantes à des lignes. Ainsi,np.arange(10, 14)qui est équivalent à[10, 11, 12, 13]correspond à la première ligne du Dataframe.
Le même Dataframe peut aussi être créé à partir des valeurs fournies en colonnes sous la forme d'un dictionnaire :
data = {"a": np.arange(10, 40, 10),
"b": np.arange(11, 40, 10),
"c": np.arange(12, 40, 10),
"d": np.arange(13, 40, 10)}
df = pd.DataFrame(data)
df.index = ["chat", "singe", "souris"]
df
- Lignes 1 à 4. Le dictionnaire
datacontient les données en colonnes. La clé associée à chaque colonne est le nom de la colonne. - Ligne 5. Le dataframe est créé avec la fonction
pd.DataFrame()à laquelle on passedataen argument. - Ligne 6. On peut définir les étiquettes des lignes de n'importe quel dataframe avec l'attribut
df.index.
22.4.2 Quelques propriétés¶
Les dimensions d'un dataframe sont données par l'attribut .shape :
Ici, le dataframe df possède trois lignes et quatre colonnes.
L'attribut .columns renvoie le nom des colonnes et permet aussi de renommer
les colonnes d'un dataframe :
La méthode .head(n) renvoie les n premières lignes du Dataframe
(par défaut, n vaut 5) :
Les Dataframes utilisés ici comme exemples sont volontairement petits. Si vous êtes confrontés à des Dataframes de grande taille, ceux-ci seront affichés partiellement dans un notebook Jupyter. Des ascenseurs en bas et à droite du Dataframe permettront de naviguer dans les données.
22.4.3 Sélections¶
Les mécanismes de sélection fournis avec pandas sont très puissants. En voici un rapide aperçu :
Sélection de colonnes¶
On peut sélectionner une colonne par son étiquette :
La notation df["Lyon"] sélectionne une colonne et renvoie un objet Series :
On trouve parfois l'écriture df.Lyon pour sélectionner une colonne. C'est une très mauvaise pratique, car cette écriture peut être confondue avec un attribut de l'objet df (par exemple .shape). Par ailleurs, elle ne fonctionne pas pour des noms de colonnes qui contiennent des espaces ou des caractères spéciaux (ce qui n'est pas non plus une bonne pratique).
Nous vous conseillons de toujours utiliser la notation df["nom_de_colonne"].
Pour sélectionner plusieurs colonnes, il faut fournir une liste de noms de colonnes :
On obtient cette fois un Dataframe avec les colonnes sélectionnées :
La sélection de plusieurs colonnes nécessite une liste entre les crochets,
par exemple df[["Lyon", "Pau"]].
Si on utilise un tuple du type df[("Lyon", "Pau")],
Python renvoie une erreur KeyError: ('Lyon', 'Pau').
Sélection de lignes¶
Pour sélectionner une ligne, il faut utiliser l'instruction .loc
et l'étiquette de la ligne :
Ici aussi, on peut sélectionner plusieurs lignes :
Enfin, on peut aussi sélectionner des lignes avec l'instruction .iloc
et l'indice de la ligne (la première ligne ayant l'indice 0) :
On peut également utiliser les tranches (comme pour les listes) :
Sélection sur les lignes et les colonnes¶
On peut bien sûr combiner les deux types de sélection (en ligne et en colonne) :
Notez qu'à partir du moment où on souhaite effectuer une sélection sur des lignes,
il faut utiliser .loc (ou .iloc si on utilise les indices).
Sélection par condition¶
Remémorons-nous d'abord le contenu du dataframe df :
Sélectionnons maintenant toutes les lignes pour lesquelles les effectifs à Pau sont supérieurs à 15 :
De cette sélection, on ne souhaite garder que les valeurs pour Lyon :
On peut aussi combiner plusieurs conditions avec & pour l'opérateur et :
et | pour l'opérateur ou :
22.4.4 Combinaison de dataframes¶
En biologie, on a souvent besoin de combiner deux tableaux à partir d'une colonne commune. Par exemple, si on considère les deux dataframes suivants :
data1 = {"Lyon": [10, 23, 17], "Paris": [3, 15, 20]}
df1 = pd.DataFrame.from_dict(data1)
df1.index = ["chat", "singe", "souris"]
df1
et
data2 = {"Nantes": [3, 9, 14], "Strasbourg": [5, 10, 8]}
df2 = pd.DataFrame.from_dict(data2)
df2.index = ["chat", "souris", "lapin"]
df2
On souhaite combiner ces deux dataframes, c'est-à-dire connaître pour les quatre villes (Lyon, Paris, Nantes et Strasbourg) le nombre d'animaux. On remarque d'ores et déjà qu'il y a des singes à Lyon et Paris, mais pas de lapin et qu'il y a des lapins à Nantes et Strasbourg, mais pas de singe. Nous allons voir comment gérer cette situation.
Pandas propose pour cela la fonction concat(), qui prend comme argument une liste de dataframes :
Lyon Nantes Paris Strasbourg
chat 10.0 NaN 3.0 NaN
singe 23.0 NaN 15.0 NaN
souris 17.0 NaN 20.0 NaN
chat NaN 3.0 NaN 5.0
souris NaN 9.0 NaN 10.0
lapin NaN 14.0 NaN 8.0
Ici, NaN indique des valeurs manquantes, cela signifie littéralement Not a Number. Mais le résultat obtenu n'est pas celui que nous attendions, puisque les lignes de deux dataframes ont été recopiées.
L'argument supplémentaire axis=1 produit le résultat attendu :
Lyon Paris Nantes Strasbourg
chat 10.0 3.0 3.0 5.0
lapin NaN NaN 14.0 8.0
singe 23.0 15.0 NaN NaN
souris 17.0 20.0 9.0 10.0
Par défaut, pandas va conserver le plus de lignes possible. Si on ne souhaite conserver que les lignes communes aux deux dataframes, il faut ajouter l'argument join="inner" :
Un autre comportement par défaut de concat() est que cette fonction va combiner les dataframes en se basant sur leurs index. Il est néanmoins possible de préciser, pour chaque dataframe, le nom de la colonne qui sera utilisée comme référence avec l'argument join_axes.
22.4.5 Opérations vectorielles¶
Pour cette rubrique, créons un Dataframe composé de nombres aléatoires compris entre 100 et 200, répartis en trois colonnes (a, b et c) et 1 000 lignes :
import numpy as np
import pandas as pd
nb_rows = 1000
df = pd.DataFrame(
{
"a": np.random.randint(100, 200, nb_rows),
"b": np.random.randint(100, 200, nb_rows),
"c": np.random.randint(100, 200, nb_rows),
}
)
Vérifions que ce Dataframe a bien les propriétés attendues :
On souhaite maintenant créer une nouvelle colonne (d) qui sera le résultat de la multiplication des colonnes a et b, à laquelle on ajoute ensuite la colonne c.
Une première manière de faire est de procéder ligne par ligne. La méthode .iterrows() permet de parcourir les lignes d'un Dataframe et renvoie un tuple contenant l'indice de la ligne (sous la forme d'un entier) et la ligne elle-même (sous la forme d'une Series) :
Ici, l'instruction .at ajoute une cellule à la ligne d'indice idx et de colonne d. Cette instruction est plus efficace que .loc pour ajouter une cellule à un Dataframe.
L'approche précédente produit le résultat attendu, mais elle n'est pas optimale, car très lente. Pour évaluer le temps moyen pour réaliser ces opérations, on utilise la commande magique %%timeit abordée dans le chapitre 18 Jupyter et ses notebooks :
qui renvoie :
Cette cellule de code s'exécute en moyenne en 52,4 ms.
Une autre approche, plus efficace, consiste à réaliser les opérations directement sur les colonnes (et non plus ligne par ligne) :
qui renvoie :
Ici, la cellule de code s'exécute en moyenne en 250 µs, soit environ 200 fois (\(52400/250\)) plus rapidement qu'avec .iterrows(). Tout comme avec les arrays du chapitre 20 Numpy, les opérations vectorielles avec les Dataframes sont rapides et efficaces. Privilégiez toujours ce type d'approche avec les arrays de NumPy ou les Series et Dataframes de pandas.
Dans l'exemple précédent, l'utilisation de la commande magique %%timeit calcule le temps d'exécution moyen d'une cellule. Python détermine automatiquement le nombre d'itérations à réaliser pour que le calcul se fasse dans un temps raisonnable. Ainsi, pour la méthode .iterrows(), le calcul est réalisé 10 fois sur sept répétitions alors que pour les opérations vectorielles, le calcul est effectué 1000 fois sur sept répétitions.
22.5 Un exemple plus concret avec les kinases¶
Pour illustrer les possibilités de pandas, voici un exemple plus concret sur un jeu de données de kinases. Les kinases sont des protéines responsables de la phosphorylation d'autres protéines.
Le fichier kinases.csv que vous pouvez télécharger
en ligne
contient des informations tirées de la base de données de séquences UniProt pour quelques kinases.
Si vous n'êtes pas familier avec le format de fichier .csv, nous vous conseillons
de consulter l'annexe A Quelques formats de données en biologie.
Avant de nous lancer dans l'analyse de ce fichier, nous vous proposons cette petite devinette :
Qu'est-ce qu'une protéine dans une piscine ?
La réponse sera donnée à la fin de ce chapitre.
22.5.1 Prise de contact avec le jeu de données¶
Une fonctionnalité très intéressante de pandas est d'ouvrir très facilement
un fichier au format .csv :
Le contenu est chargé sous la forme d'un Dataframe dans la variable df.
Le fichier contient 1 442 lignes de données plus une ligne d'en-tête. Cette dernière est automatiquement utilisée par pandas pour nommer les différentes colonnes. Voici un aperçu des premières lignes :
Entry Organism Length Creation date Mass PDB
0 A0A0B4J2F2 Human 783 2018-06-20 84930 NaN
1 A4L9P5 Rat 1211 2007-07-24 130801 NaN
2 A0A1D6E0S8 Maize 856 2023-05-03 93153 NaN
3 A0A8I5ZNK2 Rat 528 2023-09-13 58360 NaN
4 A1Z7T0 Fruit fly 1190 2012-01-25 131791 NaN
Nous avons six colonnes de données :
- l'identifiant de la protéine (
Entry) ; - l'organisme d'où provient cette protéine (
Organism) ; - le nombre d'acides aminés qui constituent la protéine (
Length) ; - la date à laquelle cette protéine a été déréférencée dans UniProt (
Creation date) ; - la masse de la protéine (
Mass), exprimée en Dalton ; - les éventuelles structures 3D de la protéine (
PDB).
La colonne d'entiers tout à gauche est un index automatiquement créé par pandas.
Nous pouvons demander à pandas d'utiliser une colonne particulière comme index.
On utilise pour cela le paramètre index_col de la fonction read_csv().
Ici, la colonne Entry s'y prête très bien, car cette colonne ne contient que
des identifiants uniques :
Organism Length Creation date Mass PDB
Entry
A0A0B4J2F2 Human 783 2018-06-20 84930 NaN
A4L9P5 Rat 1211 2007-07-24 130801 NaN
A0A1D6E0S8 Maize 856 2023-05-03 93153 NaN
A0A8I5ZNK2 Rat 528 2023-09-13 58360 NaN
A1Z7T0 Fruit fly 1190 2012-01-25 131791 NaN
La fonction .read_csv() permet également d'ouvrir un fichier au format TSV (voir l'annexe A Quelques formats de données en biologie). Il faut pour cela préciser que le séparateur des colonnes de données est une tabulation (\t), avec l'argument sep="\t".
Avant d'analyser un jeu de données, il est intéressant de l'explorer un peu. Par exemple, connaître ses dimensions :
Notre jeu de données contient donc 1 442 lignes et 5 colonnes. En effet,
la colonne Entry est maintenant utilisée comme index et n'est donc plus
prise en compte.
Il est aussi intéressant de savoir de quel type de données est constituée chaque colonne :
Les colonnes Length et Mass contiennent des valeurs numériques,
en l'occurrence des entiers (int64).
Le type object est un type par défaut.
La méthode .info() permet d'aller un peu plus loin dans l'exploration du jeu de données
en combinant les informations produites par les propriétés .shape et .dtypes :
<class 'pandas.core.frame.DataFrame'>
Index: 1442 entries, A0A0B4J2F2 to Q5F361
Data columns (total 5 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Organism 1442 non-null object
1 Length 1442 non-null int64
2 Creation date 1442 non-null object
3 Mass 1442 non-null int64
4 PDB 488 non-null object
dtypes: int64(2), object(3)
memory usage: 67.6+ KB
Avec l'argument memory_usage="deep", la méthode .info() permet de connaitre avec précision
la quantité de mémoire vive occupée par le Dataframe :
<class 'pandas.core.frame.DataFrame'>
Index: 1442 entries, A0A0B4J2F2 to Q5F361
Data columns (total 5 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Organism 1442 non-null object
1 Length 1442 non-null int64
2 Creation date 1442 non-null object
3 Mass 1442 non-null int64
4 PDB 488 non-null object
dtypes: int64(2), object(3)
memory usage: 351.0 KB
Ici, le Dataframe occupe 351 kilo-octets (ko) en mémoire.
22.5.2 Recherche de valeurs manquantes¶
Il est aussi utile de savoir si des valeurs manquantes sont présentes dans le jeu de données. Ces valeurs manquantes correspondent à des champs pour lesquels aucune valeur n'ont été fournies. Elles sont souvent représentées par NaN (pour Not a Number).
La méthode .isna() renvoie un Dataframe de la même dimension que le Dataframe initial, mais avec des valeurs booléennes (True si la valeur est manquante (NaN) ou False sinon). En le combinant avec la méthode .sum(), on peut compter le nombre de valeurs manquantes pour chaque colonne :
Ici, la seule colonne qui contient des valeurs manquantes est la colonne PDB, qui contient 954 valeurs manquantes. Cela signifie que pour 954 protéines, aucune structure 3D n'est disponible. Nous reviendrons plus tard sur cette colonne PDB.
22.5.3 Conversion en date¶
Le type object correspond la plupart du temps à des chaînes de caractères.
C'est tout à fait légitime pour la colonne Organism. Mais on sait par contre
que la colonne Creation date est une date sous la forme
année-mois-jour.
Si le format de date utilisé est homogène sur tout le jeu de données et non ambigu,
on peut demander à pandas de considérer la colonne Creation Date comme une date.
pandas détectera alors automatiquement le format de date utilisé :
L'affichage des données n'est pas modifié :
Organism Length Creation date Mass PDB
Entry
A0A0B4J2F2 Human 783 2018-06-20 84930 NaN
A4L9P5 Rat 1211 2007-07-24 130801 NaN
A0A1D6E0S8 Maize 856 2023-05-03 93153 NaN
A0A8I5ZNK2 Rat 528 2023-09-13 58360 NaN
A1Z7T0 Fruit fly 1190 2012-01-25 131791 NaN
Mais le type de données de la colonne Creation date est maintenant une date (datetime64[ns]) :
22.5.4 Statistiques descriptives et table de comptage¶
Pour les colonnes qui contiennent des données numériques, on peut obtenir
rapidement quelques statistiques descriptives avec la méthode .describe() :
Length Creation date Mass
count 1442.000000 1442 1442.000000
mean 756.139390 2001-01-25 16:10:39.112344064 84710.753814
min 81.000000 1986-07-21 00:00:00 9405.000000
25% 476.250000 1996-10-01 00:00:00 54059.000000
50% 632.000000 2002-03-10 00:00:00 71613.000000
75% 949.250000 2005-11-22 00:00:00 105485.250000
max 2986.000000 2023-09-13 00:00:00 340261.000000
std 404.195273 NaN 44764.273097
On apprend ainsi que la taille de la protéine (colonne Length)
a une valeur moyenne de 756,14 acides aminés et que
la plus petite protéine est composée de 81 acides aminés et la plus grande de 2 986. Pratique !
Des statistiques sont également proposées pour la colonne Creation date. La protéine la plus récente a ainsi été référencée le 13 septembre 2023.
La colonne Organism contient des chaînes de caractères, on peut rapidement
déterminer le nombre de protéines pour chaque organisme :
Organism
Human 489
Mouse 489
Rat 253
Fruit fly 103
Chicken 75
Rabbit 25
Maize 8
Name: count, dtype: int64
On apprend ainsi que 489 protéines sont d'origine humaine (Human) et 8 proviennent
du maïs (Maize).
22.5.5 Statistiques par groupe¶
On peut aussi déterminer, pour chaque organisme, la taille et la masse moyenne des kinases :
Length Mass
Organism
Chicken 720.160000 81120.880000
Fruit fly 784.844660 88154.669903
Human 771.004090 86281.190184
Maize 666.875000 73635.000000
Mouse 768.092025 85942.274029
Rabbit 591.480000 66754.200000
Rat 722.379447 81081.822134
La méthode .groupby() rassemble d'abord les données suivant la colonne
Organism. Puis on sélectionne les colonnes Length et Mass.
Enfin, la méthode .mean() calcule la moyenne pour chaque groupe.
Si on souhaite obtenir deux statistiques (par exemple les valeurs minimale et maximale)
en une seule fois, il convient alors d'utiliser la méthode .pivot_table(), méthode plus complexe, mais aussi beaucoup plus puissante :
min max
Length Mass Length Mass
Organism
Chicken 303 34688 2311 260961
Fruit fly 294 33180 2554 287025
Human 253 28160 2986 340261
Maize 294 33834 996 105988
Mouse 244 27394 2964 337000
Rabbit 81 9405 1382 158347
Rat 274 31162 2959 336587
- L'argument
indexprécise la colonne dont on veut agréger les données. - L'argument
valuesindique sur quelles colonnes les statistiques sont calculées. - Enfin,
aggfuncliste les statistiques calculées, ici les valeurs minimale et maximale.
Notez que les valeurs renvoyées sont d'abord les valeurs minimales pour Length
et Mass puis les valeurs maximales pour Length et Mass.
22.5.6 Analyse de données numériques¶
On peut, sans trop de risque, émettre l'hypothèse que plus il y a d'acides aminés dans la protéine, plus sa masse va être élevée.
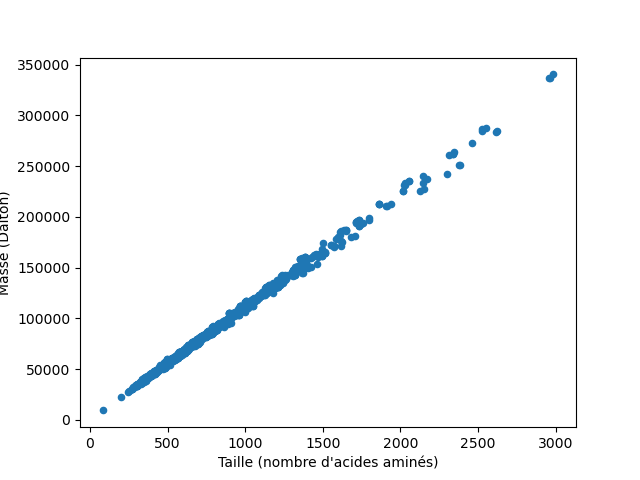
Pour vérifier cela graphiquement, on représente la masse de la protéine en fonction de sa taille (c'est-à-dire du nombre d'acides aminés) :
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.scatter(df["Length"], df["Mass"])
ax.set_xlabel("Taille (nombre d'acides aminés)")
ax.set_ylabel("Masse (Dalton)")
fig.savefig("kinases1.png")
On obtient un graphique similaire à celui de la figure 1.
Avec pandas, on peut aussi appeler une méthode .plot() sur un Dataframe
pour obtenir une représentation graphique identique à la figure 1 :
import matplotlib.pyplot as plt
df.plot(
kind="scatter",
x="Length",
y="Mass",
xlabel="Taille (nombre d'acides aminés)",
ylabel="Masse (Dalton)"
)
plt.savefig("kinases1.png")
- Ligne 4. On spécifie le type de graphique. Ici, un nuage de points.
- Lignes 5 et 6. On précise les colonnes à utiliser pour les abscisses et les ordonnées.
Le graphique de la figure 1 met en évidence une relation linéaire entre le nombre de résidus d'une protéine et sa masse.
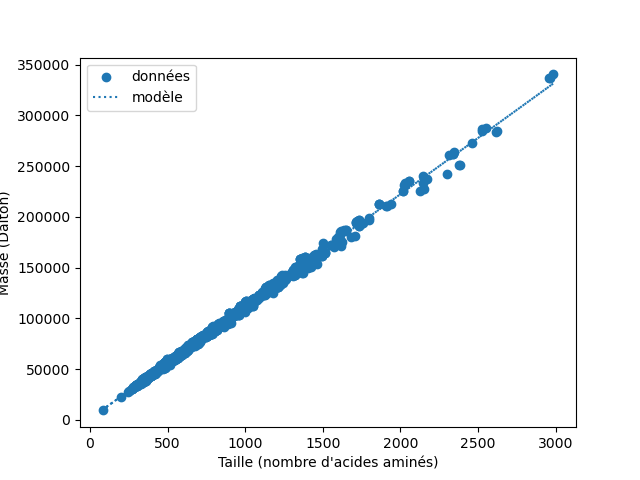
En réalisant une régression linéaire, on peut déterminer les paramètres de
la droite qui passent le plus proche possible des points du graphique.
On utilise pour cela la fonction linregress() du module scipy.stats :
LinregressResult(slope=110.63478918698122, intercept=1055.431834679228,
rvalue=0.9989676084416755, pvalue=0.0, stderr=0.13258187632073232,
intercept_stderr=113.66584551734655)
Ce modèle linéaire nous indique qu'un résidu a une masse d'environ 111 Dalton, ce qui est cohérent. On peut également comparer ce modèle aux différentes protéines :
fig, ax = plt.subplots()
ax.scatter(df["Length"], df["Mass"], label="données")
ax.plot(
df["Length"],
df["Length"]*model.slope + model.intercept,
ls=":",
label="modèle"
)
ax.set_xlabel("Taille (nombre d'acides aminés)")
ax.set_ylabel("Masse (Dalton)")
ax.legend()
fig.savefig("kinases2.png")
On obtient ainsi le graphique de la figure 2.
22.5.7 Analyse de données temporelles¶
Il peut être intéressant de savoir, pour chaque organisme, quand les premières et les dernières séquences de kinases ont été référencées dans UniProt.
La méthode .pivot_table() apporte des éléments de réponse :
min max
Creation date Creation date
Organism
Chicken 1986-07-21 2021-02-10
Fruit fly 1986-07-21 2023-09-13
Human 1986-07-21 2018-06-20
Maize 1990-08-01 2023-05-03
Mouse 1986-07-21 2017-03-15
Rabbit 1986-07-21 2010-03-02
Rat 1986-07-21 2023-09-13
Chez le poulet (Chicken), la première séquence a été référencée le 21 juillet 1986 et la dernière le 10 février 2021.
Une autre question est de savoir combien de kinases ont été référencées en fonction du temps.
La méthode .value_counts() peut être utilisée, mais elle ne renvoie que
le nombre de protéines référencées dans UniProt pour un jour donné. Par exemple,
40 structures ont été référencées le 28 novembre 2006 :
Creation date
1997-11-01 72
1996-10-01 58
2000-12-01 43
2000-05-30 41
2006-11-28 40
Name: count, dtype: int64
Si on souhaite une réponse plus globale, par exemple à l'échelle de
l'année, la méthode .resample() calcule le nombre de protéines référencées par
an (en fournissant l'argument YE). En utilisant le method chaining
présenté dans le chapitre 11 Plus sur les chaînes de caractères,
nous pouvons écrire toutes ces transformations en une seule instruction,
répartie sur plusieurs lignes pour plus de lisibilité (en utilisant des parenthèses) :
Creation date
1986-12-31 11
1987-12-31 12
1988-12-31 32
1989-12-31 29
1990-12-31 40
Freq: YE-DEC, Name: count, dtype: int64
Les dates apparaissent maintenant comme le dernier jour de l'année (31 décembre), mais désignent bien l'année complète. Dans cet exemple, 11 kinases ont été référencées dans UniProt entre le 1er janvier et le 31 décembre 1986.
Pour connaître en quelle année le plus de kinases ont été référencées dans UniProt,
il faut trier les valeurs obtenues du plus grand au plus petit avec la méthode
.sort_values(). Comme on ne veut connaître que les premières dates
(celles où il y a eu le plus de protéines référencées),
on utilisera également la méthode .head() :
Creation date
2006-12-31 167
2005-12-31 136
2004-12-31 118
2003-12-31 104
2007-12-31 88
Name: count, dtype: int64
En 2006, 167 kinases ont été référencées dans UniProt. La deuxième « meilleure » année est 2005 avec 136 protéines.
Toutes ces méthodes, enchaînées les unes à la suite des autres, peuvent vous sembler complexes, mais chacune d'elles correspond à une étape du traitement des données. Bien sûr, on aurait pu créer des variables intermédiaires pour chaque étape, mais cela aurait été plus lourd :
date1 = df["Creation date"].value_counts()
date2 = date1.resample("YE")
date3 = date2.sum()
date4 = date3.sort_values(ascending=False)
date4.head()
On aurait obtenu exactement le même résultat.
Le method chaining est une manière efficace et élégante de traiter des données avec pandas.
Enfin, pour obtenir un graphique de l'évolution du nombre de kinases référencées dans UniProt en fonction du temps, on peut encore utiliser le method chaining :
import matplotlib.pyplot as plt
(df["Creation date"]
.value_counts()
.resample("YE")
.sum()
.plot()
)
plt.savefig("kinases3.png")
On obtient ainsi le graphique de la figure 3.
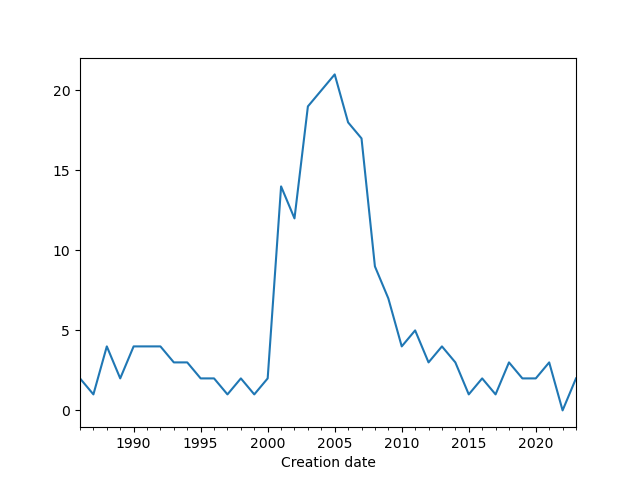
On observe un pic du nombre de kinases référencées dans UniProt sur la période 2003-2007.
22.5.8 Transformation d'une colonne¶
Nous avons vu précédemment que la colonne PDB contenait de nombreuses valeurs manquantes (NaN).
Toutefois, il est intéressant de savoir ce que peut contenir cette colonne quand elle n'est pas vide :
Organism Length Creation date Mass PDB
Entry
A2CG49 Mouse 2964 2007-10-23 337000 1WFW;7UR2;
D3ZMK9 Rat 1368 2018-07-18 147716 6EWX;
O00141 Human 431 1998-12-15 48942 2R5T;3HDM;3HDN;7PUE;
- Ligne 2. La méthode
isna()sélectionne les lignes qui contiennent des valeurs manquantes dans la colonnePDB, puis l'opérateur~inverse cette sélection. - Ligne 3. On limite l'affichage aux trois premières lignes.
On découvre que la colonne PDB contient des identifiants de structures 3D de protéines. Ces identifiants sont séparés par des points-virgules, y compris pour la dernière valeur.
Nous souhaitons compter le nombre de structures 3D pour chaque protéine. Pour cela, nous allons d'abord créer une fonction qui compte le nombre de points-virgules dans une chaîne de caractères :
Dans la ligne 2, la méthode .isna() teste si la valeur est manquante et si ce n'est pas le cas, la fonction renvoie le nombre de points-virgules dans la chaîne de caractères de la colonne PDB (ligne 5).
On applique ensuite la fonction count_structures() au Dataframe avec la méthode .apply().
On crée la nouvelle colonne nb_structures en même temps :
Organism Length Creation date Mass PDB nb_structures
Entry
A0A0B4J2F2 Human 783 2018-06-20 84930 NaN 0
A4L9P5 Rat 1211 2007-07-24 130801 NaN 0
A0A1D6E0S8 Maize 856 2023-05-03 93153 NaN 0
A0A8I5ZNK2 Rat 528 2023-09-13 58360 NaN 0
A1Z7T0 Fruit fly 1190 2012-01-25 131791 NaN 0
Les premières lignes ne sont pas très intéressantes, car elles ne contiennent pas de structures 3D. Mais on peut chercher les kinases qui ont le plus de structures 3D :
(df
.sort_values(by="nb_structures", ascending=False)
.filter(["Organism", "nb_structures"])
.head()
)
- Ligne 2. On trie les données par ordre décroissant de la colonne
nb_structures. - Ligne 3. On ne conserve que les colonnes
Organismetnb_structuresà afficher. - Ligne 4. On limite l'affichage aux cinq premières lignes.
Organism nb_structures
Entry
P24941 Human 453
P00533 Human 284
Q16539 Human 245
P68400 Human 238
P11309 Human 176
La kinase P24941 possède 453 structures 3D référencées dans UniProt. Les cinq kinases qui ont le plus de structures 3D sont toutes d'origine humaine.
Les ouvrages Python for Data Analysis (2022) de Wes McKinney et Effective Pandas (2021) de Matt Harrison sont d'excellentes références pour pandas.
La réponse à la devinette précédente est :
Une protéine kinase
(Une protéine qui nage... dans une piscine... Vous l'avez ?)
22.6 Exercices¶
Pour ces exercices, utilisez des notebooks Jupyter.
22.6.1 Analyse d'un jeu de données¶
Le jeu de données people.tsv contient les caractéristiques de quelques individus : prénom, sexe, taille (en cm) et âge (en années).
Par exemple :
| name | sex | size | age |
|---|---|---|---|
| simon | male | 175 | 33 |
| clara | female | 167 | 45 |
| serge | male | 181 | 44 |
| claire | female | 174 | 31 |
| ... | ... | ... | ... |
L'objectif de cet exercice est de manipuler ce jeu de données avec pandas, de sélectionner des données et d'en calculer quelques statistiques.
Si vous n'êtes pas familier avec le format de fichier .tsv, nous vous conseillons de consulter l'annexe A Quelques formats de données en biologie.
-
Chargement du jeu de données
- Téléchargez le fichier people.tsv.
- Ouvrez ce fichier avec pandas et la fonction
.read_csv(). N'oubliez pas de préciser le séparateur par défaut avec l'argumentsep="\t". Utilisez également l'argumentindex_colpour utiliser la colonnenamecomme index. - Affichez les six premières lignes du jeu de données.
- Combien de lignes contient le jeu de données ?
-
Sélections
- Déterminez la taille de Claire.
- Déterminez l'âge de Baptiste.
- Affichez, en une seule commande, l'âge de Paul et Bob.
-
Statistiques descriptives et table de comptage
- Déterminez la moyenne et la valeur minimale de la taille et l'âge des individus.
- Comptez ensuite le nombre de personnes de chaque sexe.
-
Statistiques par groupe
- Déterminez la taille et l'âge moyen chez les hommes et les femmes. Utilisez pour cela la méthode
.groupby().
- Déterminez la taille et l'âge moyen chez les hommes et les femmes. Utilisez pour cela la méthode
-
Sélections par filtre
- Déterminez combien de d'individus mesurent plus de 1,80 m.
- Quelle femme a moins de 35 ans ?
-
Sélections et statistiques
- Déterminez l'âge moyen des individus qui mesurent plus de 1,80 m.
- Déterminez la taille maximale des femmes qui ont plus de 35 ans.